
Responsables : Thierry Beguiristain (IR) et Bénédicte Sohm (IgR)
MembresThierry Beguiristain (IR),Arnaud Bianchi (IR), Clément Bojic (IgE), Claire Caillard (AI), Christine Friry (T), Corentin Galinier (AI), Hélène Le Cordier (IE), Sylvie Maggipinto (AjT), Orlane Scelsi (AI), Céline Simon (ASI), Bénédicte Sohm (IgR), Marie Zaffino (ASI)
Le pôle rassemble les compétences et les moyens techniques dédiés à l’analyse d’organismes vivants et/ou leurs activités à différents niveaux d’organisation biologiques (molécule, cellule, population, communauté, écosystème). Les méthodologies développées sont utilisées sur des organismes modèles en écotoxicologie aquatique et terrestre (poissons, moules d’eau douce, algues, plantes), sur des communautés de microorganismes aquatiques et terrestres et également sur des cultures de cellules eucaryotes.
Le panel d’analyses maitrisées est très diversifié (expression de gènes cibles, activités métaboliques, caractérisation de communautés bactériennes, détermination d’indice hydrobiologique…). L’utilisation de ces outils contribue à la compréhension du fonctionnement biologique des écosystèmes continentaux fortement anthropisés.
Le Pôle BE est localisé sur 3 sites Aiguillettes (A), Bridoux (B) et Charmois (C)
Compétences et plateaux techniques disponibles



Tests d'écotoxicité (terrestre, aquatique)
Clément Bojic (B), Christine Friry (A), Céline Simon (B)
- Cultures axéniques d'algues
- Elevages de microcrustacés (Daphnia magna), d'Hydra vulgaris, de vers (Eisenia fetida)
- Test normalisés d'écotoxicité sur Eisenia fetidia, végétaux, D. magna, Brachionus, P. subcapitata, F. mosseae, test Microtox
- Enceintes de cultures - Phytotrons (plateforme PEPLor) - Agitateurs incubateurs Innova S44i – Incubateurs MIR avec illumination LED à façon (Alpheus) - Fluorimètre Victor Nivo - Microtox 500
Analyse de paramètres physiologiques (Daphnies, Gammare, poisson zèbre)
Clément Bojic (B), Céline Simon (B),Marie Zaffino (ASI), Orlane Scelsi (AI)
- Respiration (Sondes PreSens)
- Comportement : déplacement avec et sans stimulus lumineux (Zantiks)
- Fréquence cardiaque (Microscope optique numérique et logiciel « maison »
Biomarqueurs, bioindicateurs
Marie Zaffino (B), Bénédicte Sohm (B), Claire Caillard (B), Christine Friry (A)
- Minéralisation du carbone (XStream), Microresp®
- Analyse de biomarqueurs
- Automates de spectrophotométrie: Konelab 20XTi et Gallery Plus
- Spectrofluorimètres (Xenius, SAFAS)
Expérimentateur Animalier (DU)
- Niveau soin : Clément Bojic
- Niveau réalisation : Bénédicte Sohm


Systématique Aquatique et terrestre
Orlane Scelsi (B)
- Capture in situ, tri, identification taxonomique des macroinvertébrés, indices biologiques normalisés (I2M2, IBGN)
- Dispositifs de piégeage pour faune du sol et aquatique. Parc optique (loupes binoculaires)
Anthracologie
Orlane Scelsi (B)
- Identification taxonomique des charbons de bois
- Parc optique (loupe binoculaire, microscope optique à lumière polarisée)

Cultures microbiennes
Thierry Beguiristain (A), Marie Zaffino (B), Hélène Le Cordier (A, C), Claire Caillard (B)
- Bactéries, champignons, cultures aérobies, anaérobies, liquides, solides, microplaques, gels de silice hybrides, dénombrement, isolement
- Autoclaves - PSM type I et II - Enceintes de cultures – Chambre anoxique et système Anoxomat
Culture cellulaire
Clément Bojic (B), Claire Caillard (B), Bénédicte Sohm (B)
- Culture de lignées cellulaires eucaryote (ZF4, ZFL, MDA-kb2, RTgill-W1)
- Salle blanche (salles propres de classe ISO 6)

Biologie moléculaire, biochimie
Thierry Beguiristain (A), Bénédicte Sohm (B), Hélène Le Cordier (A, C), Arnaud Bianchi (A)
- Quantification et expression de gènes, Western blot, mesures d'activité enzymatique
- qPCR CFX Connect & Step One - Chemidoc – Nanodrop - Spectrofluorimètre SAFAS
Transcriptomique, protéomique
Bénédicte Sohm (B)
- Analyse d'expression de gènes
- Bioanalyser - Dispositif microarray Agilent - Gels 2D (Protean IEF Cell, Ettan Dalt 6)
Bioinformatique
Arnaud Bianchi (A)
- Analyses (méta)transcriptomique, (méta) génomique
- Analyses de l’expression des gènes microbiens (trimming, mapping, normalisation, expression différentielle) ; analyse des réseaux de co-expression et réseaux de co-occurence.
- Etude de la diversité bactérienne (alpha, beta et gamma), mise en place d’arbres phylogénétique…
- Développement de workflows adaptés
- Statistiques
Microscopie
Clément Bojic (B), Corentin Galinier (B)
- Observations à haute résolution en grande profondeur de champ et selon un angle libre
- Microsc. hyperspectral Cytoviva - Microsc. à fluorescence BX41 + UC90
- Microscopie DIC (contraste interférentiel différentiel) Axio imager M1
- Microsc. numérique VHX 6000 : visualisez la circulation sanguine chez un poisson zèbre
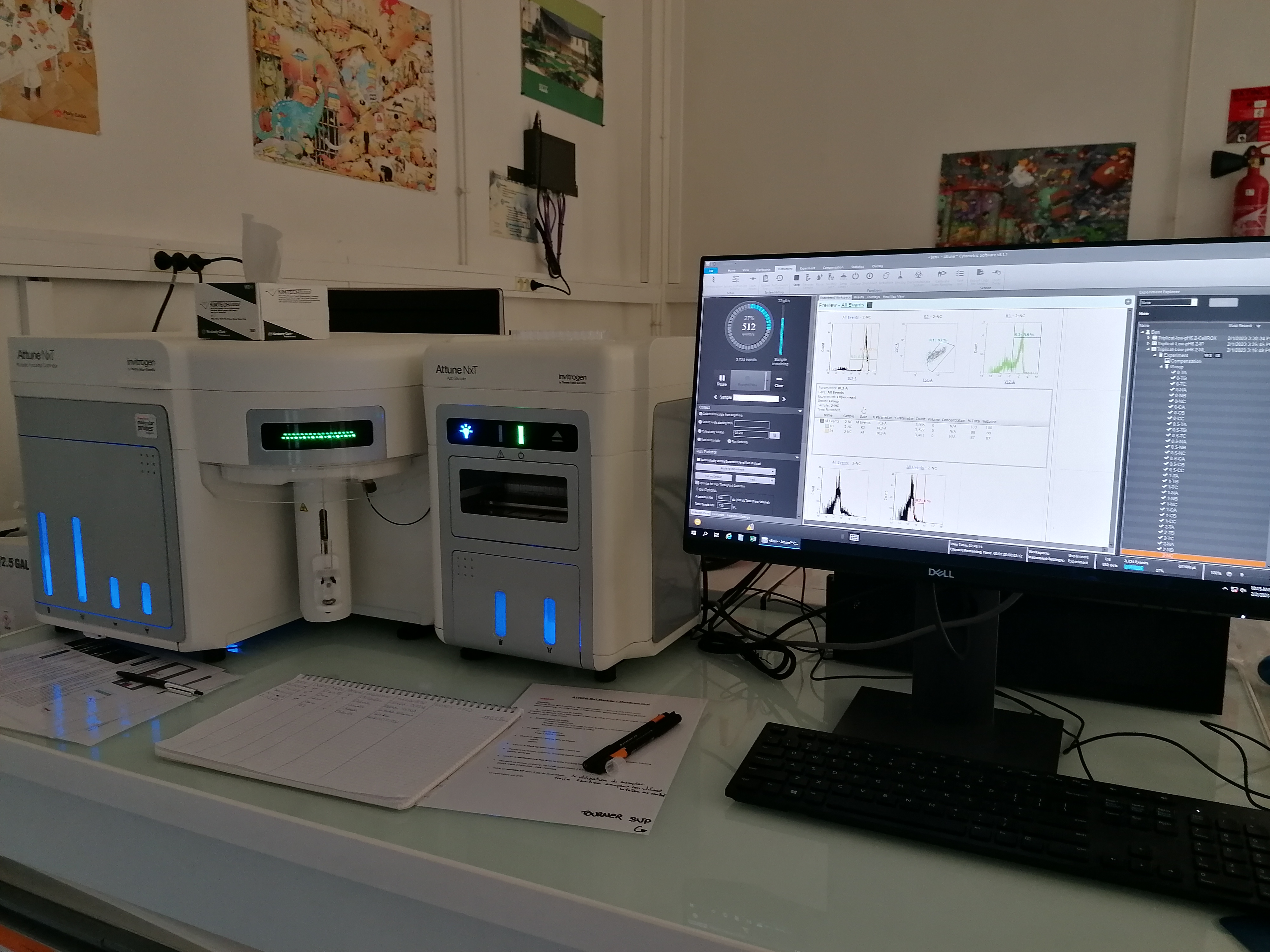
Cytomètrie
Bénédicte Sohm (B), Céline Simon (B)
- Mesure de paramètres cellulaires (viabilité, stress oxydatif, phagocytose…), marquage fluorescent de protéines
- Analyse d’interaction nanoparticules (QD, PAMAM) – cellule
- Numération algales (test normalisé, Scope For Growth)
- Attune Next (BVY – 11 colors) - Cytoflex (BR – 4 colors)
Histologie
Céline Simon (B), Claire Caillard (B)
- Coloration (H&E, Giemsa...), histochimie, détection in situ de parasites, indice gonadique de bivalves, histopathologie
- Cryostat CM1950 Leica – Ultramicrotome Ultracut 6 – Colorateur Autostainer XL

